2.1. Grundwasservorhersage mittels eines Random Forests, Hessen#
author: Noura Barakat, Melvin Hildebrand
area: Hessen
date: 26.08.2024
## Erstmal importieren wir alle benötigten libraries
import os, sys
# import netCDF4
import pandas as pd
#import datetime
#from datetime import date, datetime, timedelta
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns
import xarray as xr
import geopandas as gpd
import rioxarray
import xarray as xr
import glob
import warnings
warnings.filterwarnings("ignore", "use_inf_as_na")
---------------------------------------------------------------------------
ModuleNotFoundError Traceback (most recent call last)
Cell In[1], line 11
9 import matplotlib.pyplot as plt
10 import seaborn as sns
---> 11 import xarray as xr
12 import geopandas as gpd
13 import rioxarray
ModuleNotFoundError: No module named 'xarray'
from io import StringIO
from sklearn.ensemble import RandomForestRegressor
from sklearn.metrics import mean_squared_error
from sklearn.metrics import accuracy_score
from sklearn.model_selection import train_test_split
import math
from math import sqrt
pwd = os.getcwd()
print(pwd)
C:\Users\nunub\OneDrive\Documents\KI_2024
2.1.1. Einlesen unserer vorverarbeiteten Daten#
Wir verwenden hier bereits vorverarbeitete Daten. Der Datensatz umfasst Daten von einer Grundwassermesstelle (ID: 10319, Name: Lettgenbrunn) in Hessen. Zur Vorverarbeitung haben wir die .nc-Datei auf das AOI (Gebiet von Interesse) zugeschnitten und die .nc-Dateien für bestimmte Jahre zu einer Datei zusammengeführt. Dann haben wir Klimadaten (Temperatur- und Niederschlagswerte) an bestimmten Standorten extrahiert, sie mit GW-Pegel-Daten (Grundwasserpegel-Daten) zusammengeführt und die Daten für die Messtelle als einzelne CSV-Datei gespeichert. Diese Datei ist nun die Grundlage für den Aufbau unseres Modells.
Unser nächster Schritt ist der Modellaufbau.
data = pd.read_csv("./data/HYRAS_GWData_csv/HE_10319_weeklyData_HYRAS_GWData.csv", skipinitialspace=True).drop(['Unnamed: 0'], axis=1)
data.shape[0]
835
col2use = ['pr', 'tas', 'GWL']
#data = data.replace(np.inf, np.nan)
data.isnull().sum()
Date 0
pr 0
tas 0
GWL 148
dtype: int64
## hier werfen wir die Werte ohne Angaaben raus
data.dropna(axis=0, inplace=True)
data
| Date | pr | tas | GWL | |
|---|---|---|---|---|
| 148 | 1992-11-02 | 10.2 | 7.900000 | 371.92 |
| 149 | 1992-11-09 | 35.0 | 6.800000 | 372.46 |
| 150 | 1992-11-16 | 22.8 | 6.914286 | 373.40 |
| 151 | 1992-11-23 | 21.6 | 8.500000 | 376.69 |
| 152 | 1992-11-30 | 9.5 | 7.385714 | 376.83 |
| ... | ... | ... | ... | ... |
| 830 | 2005-11-28 | 5.9 | 2.714286 | 360.67 |
| 831 | 2005-12-05 | 7.1 | 3.157143 | 360.85 |
| 832 | 2005-12-12 | 10.2 | 3.200000 | 361.95 |
| 833 | 2005-12-19 | 12.8 | 2.514286 | 363.28 |
| 834 | 2005-12-26 | 15.3 | -0.950000 | 364.93 |
687 rows × 4 columns
sns.pairplot(data[col2use], corner=True)
plt.show()
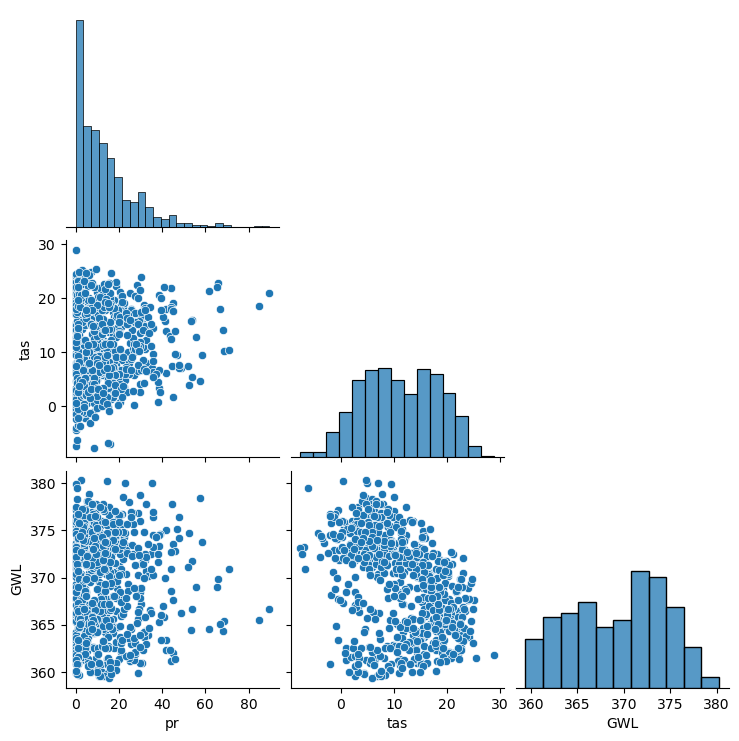
Nun integrieren wir in unseren Datensatz pro Grundwasserpegelwert die Niederschlags- (pr) und Temperaturwerte (tas) von jeweils 2, 5 und 10 Wochen im Vorhinein. Dieser Schritt ist wichtig, um die Präzision der Vorhersage zu verbessern. Der aktuelle Grundwasserpegel ist grundsätzlich auch abhängig von den Niederschlagswerten der vergangenen Wochen. Zudem kann die Temperatur der vergangenen Wochen auch ein Indikator für die Bodenfeuchte sein.
data["pr_sum_rolling_2w"] = data["pr"].rolling(2).sum()
data["pr_sum_rolling_5w"] = data["pr"].rolling(5).sum()
data["pr_sum_rolling_10w"] = data["pr"].rolling(10).sum()
data
| Date | pr | tas | GWL | pr_sum_rolling_2w | pr_sum_rolling_5w | pr_sum_rolling_10w | |
|---|---|---|---|---|---|---|---|
| 148 | 1992-11-02 | 10.2 | 7.900000 | 371.92 | NaN | NaN | NaN |
| 149 | 1992-11-09 | 35.0 | 6.800000 | 372.46 | 45.2 | NaN | NaN |
| 150 | 1992-11-16 | 22.8 | 6.914286 | 373.40 | 57.8 | NaN | NaN |
| 151 | 1992-11-23 | 21.6 | 8.500000 | 376.69 | 44.4 | NaN | NaN |
| 152 | 1992-11-30 | 9.5 | 7.385714 | 376.83 | 31.1 | 99.1 | NaN |
| ... | ... | ... | ... | ... | ... | ... | ... |
| 830 | 2005-11-28 | 5.9 | 2.714286 | 360.67 | 12.6 | 38.6 | 91.7 |
| 831 | 2005-12-05 | 7.1 | 3.157143 | 360.85 | 13.0 | 29.8 | 52.8 |
| 832 | 2005-12-12 | 10.2 | 3.200000 | 361.95 | 17.3 | 39.9 | 62.0 |
| 833 | 2005-12-19 | 12.8 | 2.514286 | 363.28 | 23.0 | 42.7 | 74.8 |
| 834 | 2005-12-26 | 15.3 | -0.950000 | 364.93 | 28.1 | 51.3 | 84.5 |
687 rows × 7 columns
data["tas_sum_rolling_2w"] = data["tas"].rolling(2).sum()
data["tas_sum_rolling_5w"] = data["tas"].rolling(5).sum()
data["tas_sum_rolling_10w"] = data["tas"].rolling(10).sum()
data
| Date | pr | tas | GWL | pr_sum_rolling_2w | pr_sum_rolling_5w | pr_sum_rolling_10w | tas_sum_rolling_2w | tas_sum_rolling_5w | tas_sum_rolling_10w | |
|---|---|---|---|---|---|---|---|---|---|---|
| 148 | 1992-11-02 | 10.2 | 7.900000 | 371.92 | NaN | NaN | NaN | NaN | NaN | NaN |
| 149 | 1992-11-09 | 35.0 | 6.800000 | 372.46 | 45.2 | NaN | NaN | 14.700000 | NaN | NaN |
| 150 | 1992-11-16 | 22.8 | 6.914286 | 373.40 | 57.8 | NaN | NaN | 13.714286 | NaN | NaN |
| 151 | 1992-11-23 | 21.6 | 8.500000 | 376.69 | 44.4 | NaN | NaN | 15.414286 | NaN | NaN |
| 152 | 1992-11-30 | 9.5 | 7.385714 | 376.83 | 31.1 | 99.1 | NaN | 15.885714 | 37.500000 | NaN |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 830 | 2005-11-28 | 5.9 | 2.714286 | 360.67 | 12.6 | 38.6 | 91.7 | 4.185714 | 26.485715 | 91.228571 |
| 831 | 2005-12-05 | 7.1 | 3.157143 | 360.85 | 13.0 | 29.8 | 52.8 | 5.871428 | 18.157143 | 80.500000 |
| 832 | 2005-12-12 | 10.2 | 3.200000 | 361.95 | 17.3 | 39.9 | 62.0 | 6.357143 | 13.842857 | 69.157143 |
| 833 | 2005-12-19 | 12.8 | 2.514286 | 363.28 | 23.0 | 42.7 | 74.8 | 5.714286 | 13.057143 | 59.800000 |
| 834 | 2005-12-26 | 15.3 | -0.950000 | 364.93 | 28.1 | 51.3 | 84.5 | 1.564286 | 10.635714 | 47.421428 |
687 rows × 10 columns
2.1.2. Was ist ein Random Forest?#
Ein Random Forest ist ein Ensemble-Lernverfahren, das aus vielen Entscheidungsbäumen besteht, die während des Trainings erstellt werden. Jeder Baum wird aus einem zufälligen Teil der Trainingsdaten und zufällig ausgewählten Features gebildet. Die Vorhersagen der einzelnen Bäume werden dann zu einer Gesamtvorhersage aggregiert, was die Genauigkeit erhöht und Überanpassung (Overfitting) reduziert. Random Forests sind robust, flexibel und können sowohl für Klassifikations- als auch für Regressionsaufgaben verwendet werden.
2.1.2.1. Schritt 1: Datensatz in Trainings- and Testdaten aufteilen#
data = data.set_index(pd.DatetimeIndex(data['Date']))
del data['Date']
data
| pr | tas | GWL | pr_sum_rolling_2w | pr_sum_rolling_5w | pr_sum_rolling_10w | tas_sum_rolling_2w | tas_sum_rolling_5w | tas_sum_rolling_10w | |
|---|---|---|---|---|---|---|---|---|---|
| Date | |||||||||
| 1992-11-02 | 10.2 | 7.900000 | 371.92 | NaN | NaN | NaN | NaN | NaN | NaN |
| 1992-11-09 | 35.0 | 6.800000 | 372.46 | 45.2 | NaN | NaN | 14.700000 | NaN | NaN |
| 1992-11-16 | 22.8 | 6.914286 | 373.40 | 57.8 | NaN | NaN | 13.714286 | NaN | NaN |
| 1992-11-23 | 21.6 | 8.500000 | 376.69 | 44.4 | NaN | NaN | 15.414286 | NaN | NaN |
| 1992-11-30 | 9.5 | 7.385714 | 376.83 | 31.1 | 99.1 | NaN | 15.885714 | 37.500000 | NaN |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 2005-11-28 | 5.9 | 2.714286 | 360.67 | 12.6 | 38.6 | 91.7 | 4.185714 | 26.485715 | 91.228571 |
| 2005-12-05 | 7.1 | 3.157143 | 360.85 | 13.0 | 29.8 | 52.8 | 5.871428 | 18.157143 | 80.500000 |
| 2005-12-12 | 10.2 | 3.200000 | 361.95 | 17.3 | 39.9 | 62.0 | 6.357143 | 13.842857 | 69.157143 |
| 2005-12-19 | 12.8 | 2.514286 | 363.28 | 23.0 | 42.7 | 74.8 | 5.714286 | 13.057143 | 59.800000 |
| 2005-12-26 | 15.3 | -0.950000 | 364.93 | 28.1 | 51.3 | 84.5 | 1.564286 | 10.635714 | 47.421428 |
687 rows × 9 columns
# Features und target trennen
X = data.drop(columns=['GWL'])
y = data['GWL']
# Daten in Trainings- und Testmengen aufteilen
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=42)
2.1.2.2. Schritt 2: Initiales Modell trainieren#
# Initialwert und Anzahl der Bäume für den Random Forest festlegen
seed = 196
n_estimators = 100
# RandomForest erstellen, X und y definieren und Modell trainieren
model = RandomForestRegressor(n_estimators = n_estimators, random_state = seed, max_features = 1.0,
min_samples_split = 2, min_samples_leaf = 1, max_depth = None)
model.fit(X_train, y_train)
RandomForestRegressor(random_state=196)In a Jupyter environment, please rerun this cell to show the HTML representation or trust the notebook.
On GitHub, the HTML representation is unable to render, please try loading this page with nbviewer.org.
RandomForestRegressor(random_state=196)
Die Zeile model.fit(X_train, y_train) startet den Trainingsprozess eines Random Forest Regressors, nachdem der Initialwert (Seed) und die Anzahl der Bäume festgelegt wurden. Die Parameter n_estimators und random_state legen die Anzahl der Entscheidungsbäume und den Zufallsstartwert fest. Weitere Parameter wie max_features, min_samples_split, min_samples_leaf und max_depth steuern die Struktur und die Komplexität der einzelnen Bäume. Während des Trainingsprozesses wird jeder Baum auf einem zufälligen Teil der Trainingsdaten (X_train, y_train) aufgebaut, wobei eine zufällige Auswahl von Merkmalen für die Splits verwendet wird. Durch die Aggregation der Vorhersagen aller Bäume wird ein robustes Modell erzeugt, das eine hohe Genauigkeit bei der Vorhersage ermöglicht und gleichzeitig Überanpassung (Overfitting) reduziert.
2.1.2.3. Schritt 3: Evaluieren#
# Bestimmheitsmaß und erklärte Varianz ausgeben lassen
print(f"Coefficient of determination: {model.score(X_train, y_train)}")
print(f"% Var explained: {model.score(X_train, y_train) * 100}")
Coefficient of determination: 0.9530484749262945
% Var explained: 95.30484749262945
In diesem Codeabschnitt werden 2 wichtige Leistungskennzahlen für das Modell ausgegeben: das Bestimmtheitsmaß und die erklärte Varianz.
Zusammengefasst gibt das Bestimmtheitsmaß bzw. die erklärte Varianz Auskunft darüber, wie gut ein Modell die Streuung der Daten erklärt. Ein höheres Bestimmtheitsmaß deutet auf ein besseres Modell hin, das die Daten gut beschreibt. Unser Ergebnis von ca. 0.95 suggeriert eine hohe Vorhersagegenauigkeit.
y_predictions = model.predict(X_test)
# Import library for metrics
from sklearn.metrics import mean_squared_error, r2_score, mean_absolute_error
# Mean absolute error (MAE)
mae = mean_absolute_error(y_test, y_predictions)
# Mean squared error (MSE)
mse = mean_squared_error(y_test, y_predictions)
# R-squared scores
r2 = r2_score(y_test, y_predictions)
mae, mse, r2
(2.0546712560386524, 8.391051875257594, 0.6901583928593296)
In diesem Codeabschnitt werden Vorhersagen für die Testdaten erstellt. Diese Vorhersagen werden dann mit den tatsächlichen Werten verglichen und im Anschluss der durchschnittliche absolute Fehler, der durchschnittliche quadrierte Fehler und das Bestmmtheistmaß berechnet. Im Durchschnitt weichen die Vorhersagen des Modells um etwa 2,05 Einheiten (hier Meter ü. NN) von den tatsächlichen Werten ab. Ein R²-Wert von 0,69 bedeutet, dass etwa 69 % der Varianz in den tatsächlichen Werten durch das Modell erklärt werden.
data
| pr | tas | GWL | pr_sum_rolling_2w | pr_sum_rolling_5w | pr_sum_rolling_10w | tas_sum_rolling_2w | tas_sum_rolling_5w | tas_sum_rolling_10w | |
|---|---|---|---|---|---|---|---|---|---|
| Date | |||||||||
| 1992-11-02 | 10.2 | 7.900000 | 371.92 | NaN | NaN | NaN | NaN | NaN | NaN |
| 1992-11-09 | 35.0 | 6.800000 | 372.46 | 45.2 | NaN | NaN | 14.700000 | NaN | NaN |
| 1992-11-16 | 22.8 | 6.914286 | 373.40 | 57.8 | NaN | NaN | 13.714286 | NaN | NaN |
| 1992-11-23 | 21.6 | 8.500000 | 376.69 | 44.4 | NaN | NaN | 15.414286 | NaN | NaN |
| 1992-11-30 | 9.5 | 7.385714 | 376.83 | 31.1 | 99.1 | NaN | 15.885714 | 37.500000 | NaN |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 2005-11-28 | 5.9 | 2.714286 | 360.67 | 12.6 | 38.6 | 91.7 | 4.185714 | 26.485715 | 91.228571 |
| 2005-12-05 | 7.1 | 3.157143 | 360.85 | 13.0 | 29.8 | 52.8 | 5.871428 | 18.157143 | 80.500000 |
| 2005-12-12 | 10.2 | 3.200000 | 361.95 | 17.3 | 39.9 | 62.0 | 6.357143 | 13.842857 | 69.157143 |
| 2005-12-19 | 12.8 | 2.514286 | 363.28 | 23.0 | 42.7 | 74.8 | 5.714286 | 13.057143 | 59.800000 |
| 2005-12-26 | 15.3 | -0.950000 | 364.93 | 28.1 | 51.3 | 84.5 | 1.564286 | 10.635714 | 47.421428 |
687 rows × 9 columns
Allerdings könnten unsere Ergebnisse auch an der Auswahl der Trainings- und Testdaten liegen. Um den Einfluss dieser Aufteilung zu reduzieren, führen wir eine sogenannte k-fache Kreuzvalidierung (k-fold cross validation) durch. Das bedeutet, dass wir die Trainingsdaten in 10 gleich große Teile aufteilen, also \(k=10\) in diesem Fall. Dann trainieren wir das Modell auf neun der 10 Teile und verwenden den 10. Teil zur Validierung des Modells wie oben beschrieben. Dieser Prozess wird 10 Mal wiederholt, wobei jedes Mal ein anderer Teil zur Validierung verwendet wird. Am Ende erhalten wir 10 RMSE-Werte, die uns einen Einblick in die Validität des oben genannten Wertes geben.
from sklearn.model_selection import cross_val_score
scores = - cross_val_score(estimator=model, X=X_train, y=y_train, cv=10, scoring='neg_mean_squared_error', n_jobs=1)
print(scores)
print(np.mean(scores), np.std(scores))
[ 7.99462509 7.6769795 8.86374395 7.83255243 9.20695896 10.75050609
6.92620051 9.73670632 8.32539769 10.4834025 ]
8.779707304687633 1.1942959488195237
2.1.2.4. Schritt 4: Hyperparameter Tuning#
Hier verwenden wir Grid Search, um die besten Hyperparameter für unser Modell zu finden. Hyperparameter sind Parameter, die nicht durch das Modelltraining selbst optimiert werden, sondern im Vorhinein festgelegt werden müssen. Sie steuern die Lernprozesse und die Architektur des Modells. Im Gegensatz zu Modellparametern, die während des Trainings durch Optimierungsalgorithmen angepasst werden, werden Hyperparameter manuell oder automatisiert (z. B. durch Grid Search) bestimmt. Grid Search durchsucht systematisch einen vordefinierten Bereich von Hyperparametern und evaluiert die Modellleistung für jede Kombination. Dies ermöglicht es, die optimalen Hyperparameter auszuwählen, die die Modellleistung maximieren und somit zu besseren Vorhersagen führen.
2.1.2.4.1. gridSearchCV#
from sklearn.model_selection import GridSearchCV
rfc = RandomForestRegressor(random_state=42)
param_grid = {
'n_estimators': [50, 100, 150, 200, 250, 300],
# 'max_features': ['auto', 'sqrt', 'log2'],
'max_depth' : [4,5,6,7,8],
# 'criterion' :['gini', 'entropy']
}
CV_rfc = GridSearchCV(estimator=rfc, param_grid=param_grid, cv= 5)
CV_rfc.fit(X_train, y_train)
GridSearchCV(cv=5, estimator=RandomForestRegressor(random_state=42),
param_grid={'max_depth': [4, 5, 6, 7, 8],
'n_estimators': [50, 100, 150, 200, 250, 300]})In a Jupyter environment, please rerun this cell to show the HTML representation or trust the notebook. On GitHub, the HTML representation is unable to render, please try loading this page with nbviewer.org.
GridSearchCV(cv=5, estimator=RandomForestRegressor(random_state=42),
param_grid={'max_depth': [4, 5, 6, 7, 8],
'n_estimators': [50, 100, 150, 200, 250, 300]})RandomForestRegressor(random_state=42)
RandomForestRegressor(random_state=42)
best_params = CV_rfc.best_params_
best_params
{'max_depth': 8, 'n_estimators': 150}
Der Hyperparameter max_depth bestimmt die maximale Tiefe der Entscheidungsbäume im Random Forest-Modell. Eine Tiefe von 8 bedeutet, dass jeder Baum im Random Forest maximal 8 Ebenen tief ist. Der Hyperparameter n_estimators gibt die Anzahl der Entscheidungsbäume im Random Forest-Modell an. In diesem Fall bedeutet n_estimators: 150, dass der Random Forest aus 150 Entscheidungsbäumen besteht.
# Initialwert für den Random Forest festlegen
seed = 196
# RandomForest erstellen, X und y definieren und Modell trainieren
model = RandomForestRegressor(n_estimators = best_params["n_estimators"],
random_state = seed,
max_features = 1.0,
min_samples_split = 2,
min_samples_leaf = 1,
max_depth = best_params["max_depth"])
model.fit(X_train, y_train)
RandomForestRegressor(max_depth=8, n_estimators=150, random_state=196)In a Jupyter environment, please rerun this cell to show the HTML representation or trust the notebook.
On GitHub, the HTML representation is unable to render, please try loading this page with nbviewer.org.
RandomForestRegressor(max_depth=8, n_estimators=150, random_state=196)
X_train
| pr | tas | pr_sum_rolling_2w | pr_sum_rolling_5w | pr_sum_rolling_10w | tas_sum_rolling_2w | tas_sum_rolling_5w | tas_sum_rolling_10w | |
|---|---|---|---|---|---|---|---|---|
| Date | ||||||||
| 1998-03-02 | 24.1 | 9.985715 | 26.400000 | 42.100000 | 95.700000 | 17.271429 | 30.771429 | 48.042858 |
| 1994-12-12 | 2.8 | 5.185714 | 8.900000 | 50.900000 | 79.000000 | 13.685714 | 38.842857 | 87.999999 |
| 1994-05-30 | 12.2 | 15.957143 | 33.100000 | 120.300000 | 196.299999 | 30.742857 | 76.385714 | 128.299999 |
| 1993-10-25 | 0.0 | 4.757142 | 20.100000 | 74.999999 | 155.799999 | 11.185713 | 47.914285 | 123.228570 |
| 1997-01-06 | 0.0 | -3.971428 | 8.700000 | 55.899998 | 169.199998 | -11.628571 | -14.571429 | 11.257143 |
| ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 1994-03-14 | 18.7 | 7.628571 | 32.300000 | 55.200000 | 98.300000 | 17.528572 | 28.728572 | 48.328572 |
| 1994-11-14 | 16.4 | 10.528571 | 25.800000 | 44.500000 | 110.500000 | 19.814285 | 49.799999 | 111.528569 |
| 1998-01-05 | 7.0 | 6.714286 | 23.400000 | 76.100000 | 145.900000 | 13.614286 | 28.142858 | 52.828572 |
| 2001-03-05 | 10.6 | 8.071428 | 21.899999 | 49.299999 | 122.699997 | 11.242857 | 28.728571 | 44.385714 |
| 1994-10-17 | 3.2 | 8.685714 | 3.200000 | 35.800000 | 152.700000 | 18.571428 | 56.799998 | 140.028566 |
549 rows × 8 columns
2.1.2.5. Schritt 5: Plotten#
stats = model.get_params()
feature_importance = model.feature_importances_
features = X.columns
var_imp_df = pd.DataFrame({'Feature': features, 'Importance': feature_importance})
var_imp_df = var_imp_df.sort_values(by = 'Importance', ascending = False).head(54)
var_imp_10 = var_imp_df.head(10)
# Plotten der wichtigsten Variablen
plt.figure(figsize = (8, 3))
sns.barplot(x = 'Importance', y = 'Feature', data = var_imp_10, color = 'pink')
plt.title("δ¹⁸O")
plt.xlabel("Importance score")
plt.ylabel("Feature")
plt.text(0, -1.5, f"n_estimators = {n_estimators}, max_features = {max_features}\nRMSE = {rmse:.2f}, R² = {r2:.2f}",
fontsize = 10, ha = 'left', va = 'center')
plt.show()
---------------------------------------------------------------------------
NameError Traceback (most recent call last)
Cell In[373], line 7
5 plt.xlabel("Importance score")
6 plt.ylabel("Feature")
----> 7 plt.text(0, -1.5, f"n_estimators = {n_estimators}, max_features = {max_features}\nRMSE = {rmse:.2f}, R² = {r2:.2f}",
8 fontsize = 10, ha = 'left', va = 'center')
9 plt.show()
NameError: name 'max_features' is not defined
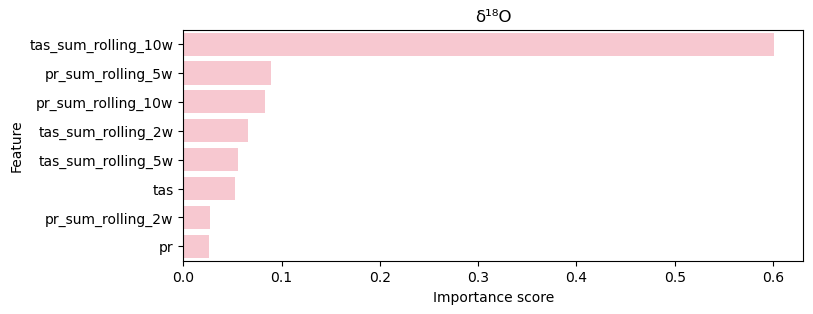
Hierbei ist klar geworden, dass die Variable der Temperatur 10 Wochen im Vorhinein die Wichtigste ist.
data_1992_2004 = data[(data.index.year >= 1992) & (data.index.year <= 2004)]
data_2005 = data[(data.index.year >= 2005)]
data.index.year
Index([1992, 1992, 1992, 1992, 1992, 1992, 1992, 1992, 1992, 1993,
...
2005, 2005, 2005, 2005, 2005, 2005, 2005, 2005, 2005, 2005],
dtype='int32', name='Date', length=687)
data_1992_2004
| pr | tas | GWL | pr_sum_rolling_2w | pr_sum_rolling_5w | pr_sum_rolling_10w | tas_sum_rolling_2w | tas_sum_rolling_5w | tas_sum_rolling_10w | |
|---|---|---|---|---|---|---|---|---|---|
| Date | |||||||||
| 1992-11-02 | 10.2 | 7.900000 | 371.92 | NaN | NaN | NaN | NaN | NaN | NaN |
| 1992-11-09 | 35.0 | 6.800000 | 372.46 | 45.2 | NaN | NaN | 14.700000 | NaN | NaN |
| 1992-11-16 | 22.8 | 6.914286 | 373.40 | 57.8 | NaN | NaN | 13.714286 | NaN | NaN |
| 1992-11-23 | 21.6 | 8.500000 | 376.69 | 44.4 | NaN | NaN | 15.414286 | NaN | NaN |
| 1992-11-30 | 9.5 | 7.385714 | 376.83 | 31.1 | 99.1 | NaN | 15.885714 | 37.500000 | NaN |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 2004-11-29 | 3.1 | 4.457143 | 374.78 | 7.5 | 41.0 | 120.200002 | 9.771429 | 27.157143 | 87.857143 |
| 2004-12-06 | 0.0 | 0.528571 | 372.96 | 3.1 | 36.2 | 107.200002 | 4.985714 | 17.628572 | 74.271429 |
| 2004-12-13 | 12.5 | 1.028571 | 371.89 | 12.5 | 27.7 | 99.500002 | 1.557143 | 15.457143 | 61.714286 |
| 2004-12-20 | 4.6 | 2.157143 | 371.48 | 17.1 | 24.6 | 90.500002 | 3.185714 | 13.485715 | 54.757144 |
| 2004-12-27 | 12.6 | 3.357143 | 373.36 | 17.2 | 32.8 | 98.600002 | 5.514286 | 11.528572 | 45.842858 |
635 rows × 9 columns
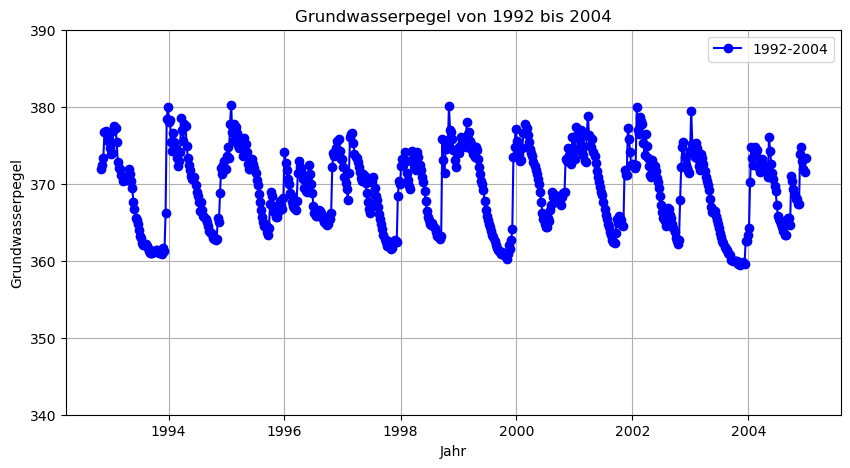
# Daten zwischen 1992 und 2004 plotten
plt.figure(figsize=(10, 5))
plt.plot(data_1992_2004.index, data_1992_2004['GWL'], marker='o', linestyle='-', color='b', label='1992-2004')
plt.xlabel('Jahr')
plt.ylabel('Grundwasserpegel')
plt.title('Grundwasserpegel von 1992 bis 2004')
plt.legend()
plt.grid(True)
plt.ylim(340, 390)
plt.show()
# Daten von 2005 plotten
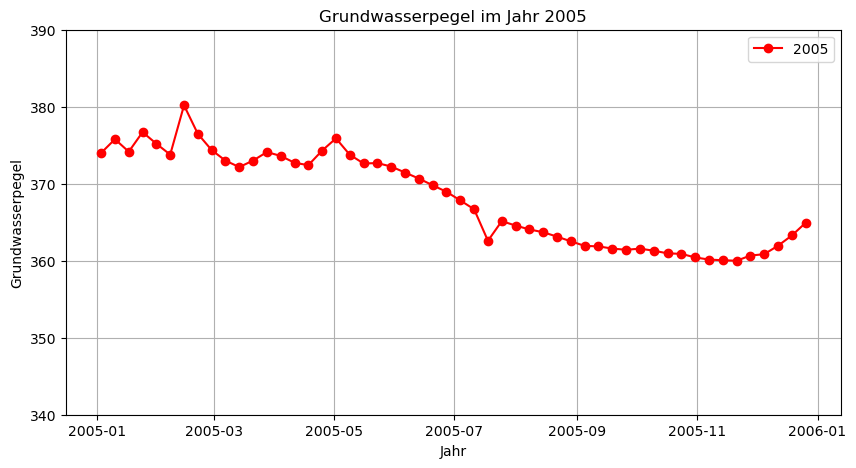
plt.figure(figsize=(10, 5))
plt.plot(data_2005.index, data_2005['GWL'], marker='o', linestyle='-', color='r', label='2005')
plt.xlabel('Jahr')
plt.ylabel('Grundwasserpegel')
plt.title('Grundwasserpegel im Jahr 2005')
plt.legend()
plt.grid(True)
plt.ylim(340, 390)
plt.show()
X_val, y_val=data_2005.drop(columns=['GWL']), data_2005['GWL']
y_predictions_2005 = model.predict(X_val)
# Die Vorhersagewerte und die tatsächlichen Werte aus 2005 gleichzeitig plotte, um Modelleistung zu sehen.
plt.plot(X_val.index, y_predictions_2005, marker='o', linestyle='-', color = 'red', label = 'Vorhersagewerte')
plt.plot(X_val.index, y_val, marker='o', linestyle='-', color = 'green', label = 'tatsächliche Werte')
plt.xlabel('Jahr')
plt.ylabel('Grundwasserpegel')
plt.title('Grundwasserpegel im Jahr 2005')
plt.legend()
plt.show()
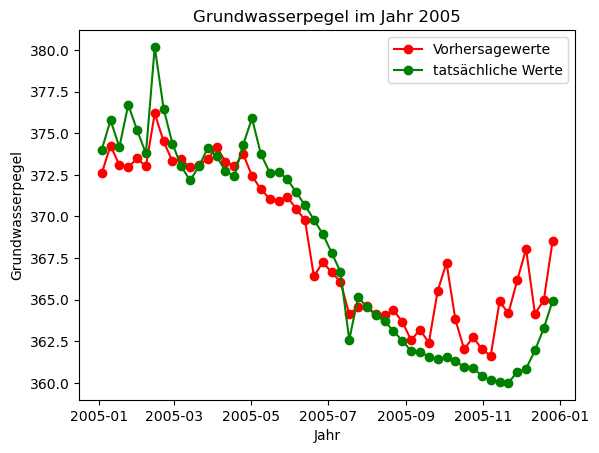
Hierbei stellt der rote Graph die vom Modell vorhergesagten Were an, wobei der grüne Graph die tatsächlichen Daten von 2005 darstellt. Wir sehen, dass die Diskrepanzen zwischen den 2 Graphen relativ klein sind.
2.1.3. Diskussion#
Zusammenfassend haben wir hier ein Random-Forest-Modell entwickelt, um die Grundwasserstände an der Messstelle Lettgenbrunn in Hessen vorherzusagen. Dafür wurden die Grundwasserdaten der Messstelle, sowie die Temperatur- und Niederschlagsdaten für den Zeitraum von 1992 bis 2005 gesammelt und in das Modell eingespeist. Die Entscheidung, die Vorhersage auf eine einzelne Messstelle zu beschränken, wurde getroffen, um Overfitting zu vermeiden. Anfangs hatten wir versucht, die Daten mehrerer Messstellen zu aggregieren, jedoch führte dies zu einer geringeren Modellleistung. Dies lag wahrscheinlich an der hohen Heterogenität der Daten, insbesondere der Niederschlags- und Temperaturwerte, die stark zwischen den verschiedenen Messstellen variierten.
Ein wesentlicher Schritt zur Verbesserung der Modellgenauigkeit war die Erweiterung der Features um historische Daten. Neben den aktuellen Temperatur- und Niederschlagswerten wurden auch die Werte von 2, 5 und 10 Wochen zuvor einbezogen. Diese Erweiterung der Features hat sich als vorteilhaft erwiesen, da Temperatur- und Niederschlagswerte wichtige Indikatoren für die Bodenfeuchte sind und historische Werte einen bedeutenden Einfluss auf den aktuellen Grundwasserstand haben. Die Integration dieser zusätzlichen Datenpunkte hat die Vorhersagegenauigkeit deutlich verbessert.
Das Modell erzielte bei den Trainingswerten ein Bestimmtheitsmaß (R²) bzw. eine erklärte Varianz von ca. 95%, was auf eine gute Modellleistung hinweist. Bei den Testdaten erreichte das Modell ein Bestimmheitsmaß von ca. 69%. Zur Validierung des Modells wurden die vorhergesagten Grundwasserstände für das Jahr 2005 mit den tatsächlichen Werten verglichen. Die simultane Darstellung beider Datensätze in einem Plot zeigte, dass die vorhergesagten Werte den tatsächlichen Werten ziemlich nahe kamen.
Trotzdem beobachten wir, dass die Vorhersage fehlerhaft ist. Besonders im Umgang mit extremen Werten, scheint das Modell Fehler zu machen. Ein möglicher Grund hierfür könnte die Komplexität von Grundwasserdaten generell und die limitierte Datenverfügbarkeit sein. Grundwasserstände werden von zahlreichen Faktoren beeinflusst, die in komplexen, nichtlinearen Beziehungen zueinander stehen. Einflussfaktoren wie die Bodenbeschaffenheit, anthropogene Einflüsse, die Landnutzung und die Vegetation wurden bei unserem Modell außer Acht gelassen. Die Intergration der genannten Einflussfaktoren in den Datensatz hätte vermutlich ein genaueres Vorhersagergebnis erzielt. Ferner neigen Random Forest-Modelle dazu, extremere Werte zu glätten, da sie auf dem Durchschnitt vieler Entscheidungsbäume basieren und extreme Ausprägungen in den Entscheidungsbäumen in den Mittelwert miteinfließen. Dies kann dazu führen, dass Extremwerte flacher vorausgesagt werden, als sie tatsächlich sind, wie das in unserem Modell bis ca. August 2005 der Fall ist. Unser Modell ist etwas überangepasst (overfittet), was durch die Diskrepanz zwischen den erklärten Varianzen der Trainings- und Testdaten gezeigt wird. Dies spiegelt sich in der teilweise Ungenauigkeit unserer Vorhersage wider. Außerdem sagt das Modell die Fluktuationen zum Teil extremer voraus, als sie eigentlich sind, z.B. zwischen September und November 2005. Dies könnte daran liegen, dass in den Trainingsdaten die Fluktuationen zum Ende des Jahres tendenziell extremer waren, also Extremwerte überrepräsentiert waren und das Modell diese Extremwerte in der Vorhersage überbetont.
Insgesamt zeigt die Modellleistung jedoch, dass der Random Forest eine leistungsfähige Methode zur Vorhersage von Grundwasserständen ist, insbesondere wenn relevante historische Daten einbezogen werden. Weitere Verfeinerungen des Modells und eine erweiterte Datengrundlage um weitere Eiflussfaktoren für Gundwasserstände könnten die Vorhersagegenauigkeit weiter verbessern.